생의학(Biomedicine)은 생물학과 의학의 원리를 적용하여 건강과 질병을 연구하는 분야입니다.
이는 인체의 생리학, 생화학, 분자생물학 등을 바탕으로 질병의 원인, 예방, 진단 및 치료 방법을 개발하는 데 중점을 둡니다.
바탕으로 질병의 원인, 예방, 진단 및 치료 방법을 개발하는 데 중점을 둡니다.Abstract
이 논문의 주요 내용을 요약하면 다음과 같습니다:
- 기존 딥러닝의 한계점 언급
- 일반적으로 딥 네트워크를 성공적으로 학습시키기 위해서는 수천 개의 레이블된 학습 데이터가 필요하다는 인식이 있었음
- 제안하는 해결책
- 데이터 증강(data augmentation)을 적극적으로 활용하여 적은 수의 학습 데이터를 효율적으로 사용하는 네트워크와 학습 전략을 제시
- 네트워크 구조의 특징
- Contracting path(수축 경로): 문맥 정보를 포착
- Symmetric expanding path(대칭적 확장 경로): 정확한 위치 파악 가능
- 두 경로가 대칭적으로 구성된 구조
- 성능 검증
- ISBI challenge에서 기존 최고 성능(sliding-window CNN)을 뛰어넘음
- 전자현미경 스택의 신경 구조 분할 작업에서 우수한 성능 달성
- 투과광 현미경 이미지에서도 2015 ISBI cell tracking challenge 우승
- 실용적 장점
- 빠른 처리 속도: 512x512 이미지 분할에 GPU에서 1초 미만 소요
- Caffe 기반 구현체와 학습된 네트워크 공개
1. Introduction


이 내용은 U-Net 개발의 배경과 핵심 아이디어를 설명합니다.
- 딥러닝의 발전과 한계
- 최근 2년간 CNN이 많은 시각 인식 작업에서 최고 성능 달성
- ImageNet의 백만 개 학습 이미지로 8층 네트워크 학습이 breakthrough
- 하지만 의생명 분야에서는:
- 픽셀별 분류(localization) 필요
- 수천 개의 학습 이미지 확보가 어려움
- 기존 해결책(Ciresan et al.)과 한계
- sliding-window 방식으로 각 픽셀 주변 패치를 입력으로 사용
- 한계점:
- 패치별 개별 처리로 인한 느린 속도
- 중복되는 패치로 인한 비효율성
- localization 정확도와 context 활용 사이의 trade-off
- U-Net의 핵심 아키텍처
- "fully convolutional network" 기반 확장
- 주요 특징:
- contracting path와 대칭적인 expanding path
- 상향 샘플링으로 해상도 증가
- 고해상도 특징과 상향 샘플링된 출력 결합
- fully connected layer 없음
- valid convolution만 사용
- 주요 기술적 혁신
- Overlap-tile 전략:
- 큰 이미지의 seamless segmentation 가능
- GPU 메모리 한계 극복
- 데이터 증강:
- elastic deformation 적용
- 적은 훈련 데이터로도 효과적 학습 가능
- Weighted loss:
- 같은 클래스의 접촉된 객체 분리 문제 해결
- 접촉된 세포 사이 배경에 큰 가중치 부여
- Overlap-tile 전략:
- 적용 및 성과
- EM 스택의 신경 구조 분할에서 Ciresan et al. 능가
- ISBI cell tracking challenge 2015의 2D 투과광 데이터셋에서 큰 격차로 우승
이 논문은 의료 영상 분할의 특수한 요구사항(적은 데이터, 정확한 위치 파악, 객체 분리 등)을 해결하는 혁신적인 아키텍처를 제안했습니다.
2. Network Architecture
이 내용은 U-Net의 구체적인 네트워크 아키텍처를 설명하는 부분입니다.
- 전체 구조의 두 가지 주요 경로
- Contracting path (좌측)
- Expansive path (우측)
- Contracting Path의 구조
- 반복되는 기본 블록:
- 두 번의 3x3 convolution (패딩 없음)
- 각 convolution 후 ReLU 활성화 함수
- 2x2 max pooling (stride=2)로 다운샘플링
- 특징:
- 다운샘플링 단계마다 feature channel 수를 2배로 증가
- 반복되는 기본 블록:
- Expansive Path의 구조
- 각 단계별 구성:
- Feature map의 upsampling
- 2x2 up-convolution (feature channel 수를 절반으로 감소)
- Contracting path의 cropped feature map과 concatenation
- 두 번의 3x3 convolution (각각 ReLU 적용)
- 각 단계별 구성:
- 최종 출력층
- 1x1 convolution 사용
- 64개 특징 벡터를 원하는 클래스 수로 매핑
- 주요 특징들
- 총 23개의 convolutional layer
- 각 convolution에서 테두리 픽셀 손실 발생
- Seamless tiling을 위해 입력 타일 크기는 2x2 max-pooling이 짝수 크기의 x, y에 적용되도록 선택
- 구현상의 중요 포인트
- 패딩 없는(unpadded) convolution 사용
- Feature map 연결 시 cropping 필요
- 입력 크기 선택이 중요 (max-pooling 고려)
이 구조는 특히 의료 영상 분할에 효과적인데, contracting path에서 문맥 정보를 포착하고 expansive path에서 정확한 위치 정보를 복원하면서, 두 경로의 feature map을 연결함으로써 세밀한 분할이 가능하게 합니다.
3. Training


이 내용은 U-Net의 학습 방법과 구현 세부사항을 설명하는 부분입니다.
- 학습 구현의 기본 사항
- Caffe 프레임워크의 확률적 경사 하강법(SGD) 사용
- 배치 크기와 입력 관련:
- 패딩 없는 convolution으로 출력 이미지가 입력보다 작음
- GPU 메모리 효율을 위해 큰 입력 타일 선호
- 배치 크기는 1로 설정
- 높은 모멘텀(0.99) 사용하여 이전 학습 샘플들의 영향력 유지
- 손실 함수(Energy Function) 설계
- Soft-max와 cross entropy 결합:
- Soft-max:

ak(x): 픽셀 위치 x에서 특징 채널 k의 활성화 K: 클래스 수 pk(x): 근사된 최대 함수 - Cross entropy:
- Soft-max:
- Soft-max와 cross entropy 결합:

- 가중치 맵(Weight Map) 설계
- 사전 계산되는 가중치 맵의 목적:
- 클래스 빈도 차이 보상
- 접촉된 세포 간 분리 경계 학습 강화
- 계산 공식:

wc(x): 클래스 빈도 균형을 위한 가중치d1(x): 가장 가까운 세포 경계까지의 거리d2(x): 두 번째로 가까운 세포 경계까지의 거리실험에서 w0 = 10, σ ≈ 5 pixels 사용
- 사전 계산되는 가중치 맵의 목적:
- 가중치 초기화의 중요성
- 깊은 네트워크에서 적절한 가중치 초기화의 중요성 강조
- 목표: 각 특징 맵이 대략적으로 unit variance를 가지도록
- 초기화 방법:
- 가우시안 분포에서 가중치 추출
- 표준편차 = √(2/N)
- N: 뉴런의 입력 노드 수
- 예: 3x3 convolution, 64 채널일 때 N = 9·64 = 576
이 설명은 U-Net의 실제 구현에 필요한 중요한 세부사항들을 다루고 있으며, 특히 가중치 맵을 통한 세포 분리 문제 해결과 효과적인 학습을 위한 구체적인 파라미터 설정을 제시합니다.
--------------------------------
- 학습 구현의 기본 사항
- Caffe의 확률적 경사 하강법(SGD) 사용
- 패딩되지 않은 합성곱으로 인해 출력 이미지가 입력보다 작음
- GPU 메모리 효율을 위해 큰 배치 크기 대신 큰 입력 타일 선호
- 배치 크기는 1로 설정
- 높은 모멘텀(0.99) 사용하여 이전 학습 샘플들의 영향력 증가
- 에너지 함수 구성
- 최종 특징 맵에 대한 픽셀별 소프트맥스와 교차 엔트로피 손실 함수 결합
- 소프트맥스 함수:
- 가중치 맵(weight map) 특징
- 특정 픽셀에 더 많은 중요도를 부여하기 위해 도입
- 각 클래스 픽셀의 빈도 차이를 보상
- 접촉된 세포 사이의 작은 분리 경계를 학습하도록 강제
- 계산 공식:
- 가중치 초기화
- 깊은 네트워크에서 가중치 초기화가 매우 중요
- 각 특징 맵이 대략적으로 단위 분산을 가지도록 설계 p 2/N
- 가우시안 분포에서 표준편차 √(2/N)로 초기 가중치 추출
- N: 하나의 뉴런에 입력되는 노드 수
- 예: 3x3 합성곱, 64 특징 채널의 경우 N = 9 · 64 = 576
이 설명은 U-Net의 학습 과정에서 사용되는 다양한 기술적 세부사항과 그 이유를 상세히 설명합니다.
3-1. Data Augmentation
이 내용은 U-Net에서 사용된 데이터 증강(Data Augmentation) 기법에 대한 설명입니다.
- 데이터 증강의 필요성
- 적은 훈련 샘플로도 네트워크가 원하는 특성을 학습하게 하기 위해 필수적
- 목표: invariance(불변성)과 robustness(견고성) 확보
- 현미경 이미지에서 필요한 불변성/견고성
- Shift invariance (이동 불변성)
- Rotation invariance (회전 불변성)
- Deformation robustness (변형에 대한 견고성)
- Gray value variations robustness (명암값 변화에 대한 견고성)
- 핵심 데이터 증강 기법: Random Elastic Deformation
- 적은 수의 주석이 달린 이미지로 학습할 때 가장 중요한 개념
- 구현 방법:
- 3x3 격자에 무작위 변위 벡터 생성
- 가우시안 분포에서 변위 샘플링 (표준편차 10 픽셀)
- 이중 3차 보간법(bicubic interpolation)으로 픽셀별 변위 계산
- 추가적인 데이터 증강
- Contracting path 끝 부분에 dropout 층 배치
- 이를 통한 암묵적 데이터 증강 효과
이 설명은 U-Net이 제한된 훈련 데이터로도 효과적인 성능을 달성할 수 있는 핵심 전략인 데이터 증강 기법을 상세히 설명합니다.
4. Experiments
이 내용은 U-Net을 세 가지 다른 세포 분할 작업에 적용한 실험 결과를 설명하는 부분입니다.
- 전자현미경 신경 구조 분할 실험
- 데이터셋: ISBI 2012 EM segmentation challenge
- 학습 데이터: 30개 이미지 (512x512 픽셀)
- 초파리 유충의 ventral nerve cord 전자현미경 이미지
- 각 이미지에 세포(흰색)와 막(검은색)이 레이블링됨
- 평가 방법:
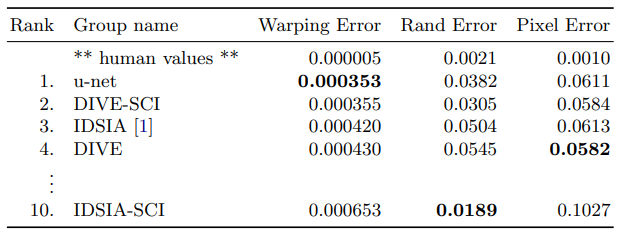
- 10개 임계값에서의 warping error, Rand error, pixel error 계산
- 결과:
- 입력 데이터의 7개 회전 버전 평균 사용
- Warping error: 0.0003529 (새로운 최고 점수)
- Rand error: 0.0382
- Ciresan et al.의 결과(warping error: 0.000420, rand error: 0.0504)보다 우수
- 데이터셋: ISBI 2012 EM segmentation challenge
- 광학현미경 세포 분할 실험
- ISBI cell tracking challenge 2014/2015의 일부
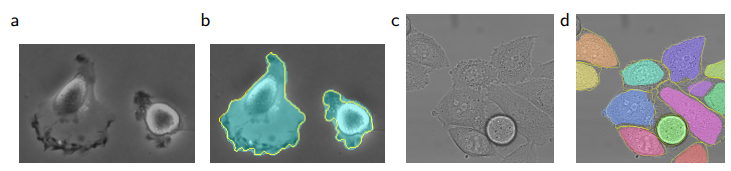
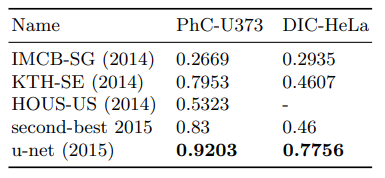
- 첫 번째 데이터셋 "PhC-U373":
- Glioblastoma-astrocytoma U373 세포
- 35개의 부분적으로 주석된 학습 이미지
- 평균 IOU: 92% (2위: 83%)
- 두 번째 데이터셋 "DIC-HeLa":
- 유리 표면의 HeLa 세포
- 20개의 부분적으로 주석된 학습 이미지
- 평균 IOU: 77.5% (2위: 46%)
5. Conclusion
이 내용은 U-Net의 전반적인 성능과 장점을 요약한 결론 부분입니다.
- U-Net의 성능
- 매우 다양한 생물의학 분할 작업에서 우수한 성능 달성
- 효율적인 학습
- elastic deformation을 이용한 데이터 증강 덕분에:
- 매우 적은 수의 주석 처리된 이미지만으로도 학습 가능
- 합리적인 학습 시간: NVidia Titan GPU(6GB)에서 10시간만 소요
- elastic deformation을 이용한 데이터 증강 덕분에:
- 구현 공유
- Caffe 기반 전체 구현체 제공
- 학습된 네트워크 모델 제공
- 확장성
- 더 많은 다양한 작업에 쉽게 적용 가능할 것으로 기대
Reference
논문 출저: U-Net: Convolutional Networks for Biomedical Image Segmentation
리디렉션 알림
www.google.com



